王秀杰研究组开发单细胞组学细胞标记基因鉴定算法COSG
发布时间:2022.01.25
在单细胞测序数据分析中,对细胞进行准确分类是数据分析的重要基础,然而如何鉴定不同细胞类型的细胞标记基因(cell marker gene)仍面临巨大的挑战。目前常用的细胞标记基因鉴定方法通常基于差异表达分析来寻找在目标细胞类群和其余细胞之间存在表达差异的基因,并以此作为目标类群的细胞标记基因。然而,基于差异表达分析的方法通常无法保证鉴定到的细胞标记基因在目标类群细胞中具有表达特异性,并且还存在耗时长等不足。随着单细胞RNA测序、单细胞ATAC测序和空间转录组测序技术的发展,也需要一种能够从不同类型测序数据中鉴定细胞标记基因的通用方法。
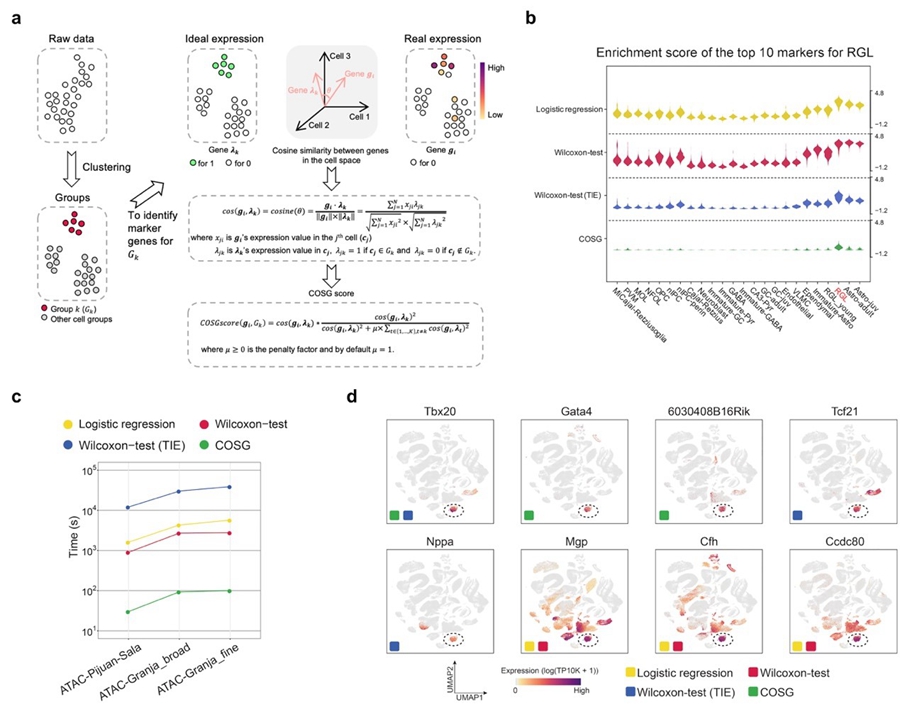
针对上述问题,中国科学院遗传与发育生物学研究所王秀杰研究组开发了一种基于余弦相似度的方法COSG,能够更加准确和快速地从单细胞测序数据中鉴定出细胞标记基因。COSG的核心思想是通过比较基因在n维细胞空间中的向量夹角来描述基因在所有细胞中表达模式的相似性。在细胞空间内,每个维度代表一个细胞,每个基因在n维细胞空间内所对应的向量由n个基(basis)组成,其中n为总细胞数。基因在每个基上的坐标代表该基因在每个细胞中的表达水平。因此,在n维细胞空间中,两个基因的余弦相似度等价于这两个基因所对应向量的夹角的余弦值。基因在所有细胞上的表达模式越相似,其对应向量的夹角就越小。如果两个基因有相同的表达模式,那么无论它们的表达丰度是否存在差异,这两个基因所对应的向量之间的夹角为零。因此,余弦相似度的计算不受基因表达丰度的影响,可以更加准确地鉴定出细胞标记基因。同时,由于单细胞测序数据具有稀疏性(包含大量零值)的特点,而余弦相似度对稀疏矩阵的计算非常高效,所以COSG在计算时间上具有很大优势。
COSG适用于单细胞RNA测序数据、单细胞ATAC测序数据和空间转录组数据。同时,COSG具有很好的计算效率和可扩展性,能应用于超过百万细胞的大规模数据集,并在不到2分钟的时间内鉴定出超过100万个细胞的标记基因。在多种模拟数据和真实数据集上的评测结果表明,COSG鉴定出的标记基因或标记基因组区域具有更好的细胞类型特异性,表明COSG相比于其他现有方法在准确性和计算效率上具有明显优势。
该研究结果以“Accurate and fast cell marker gene identification with COSG”为题于2022年1月19日在线发表在《Briefings in Bioinformatic》杂志上。COSG程序的代码已通过GitHub网站开源发布。COSG程序的Python语言版本和R语言版本的代码链接分别为https://github.com/genecell/COSG和https://github.com/genecell/COSGR,并且可以很方便地整合到常用的单细胞数据分析软件Scanpy和Seurat中。遗传发育所王秀杰研究组博士研究生戴敏为该论文第一作者,王秀杰研究员和华中科技大学软件学院裴小兵教授为论文的共同通讯作者。相关工作得到国家重点研发计划项目、国家自然科学基金委、中科院战略性先导专项、北京市自然科学基金项目的资助。
 |
|
图: COSG的计算流程和评测结果。(a) COSG的计算流程。(b) Logistic regression、Wilcoxon-test、Wilcoxon-test (TIE)和COSG四种方法鉴定得到的RGL细胞前10个标记基因的富集得分比较。(c) Logistic regression、Wilcoxon-test、Wilcoxon-test (TIE)和COSG在三个不同单细胞ATAC测序数据集上的运行时间比较。(d) Logistic regression、Wilcoxon-test、Wilcoxon-test (TIE)和COSG鉴定得到的心脏成纤维细胞的前3个标记基因的表达模式。 |