曹晓风研究组在拟南芥精氨酸甲基转移酶的功能研究中取得新进展
发布时间:2016.04.27
蛋白质翻译后修饰是调节蛋白质生物学功能的关键步骤,目前已发现的蛋白质翻译后修饰形式多达百种以上。其中精氨酸甲基化是真核生物中广泛存在且进化上保守的翻译后修饰,对蛋白质生物学功能具有十分特殊的调节作用,参与调控种重要的生物学过程。蛋白质精氨酸甲基转移酶(PRMT)负责催化精氨酸甲基化,动物中PRMT的缺失突变不仅会导致严重的生长发育异常,而且与人类遗传疾病与癌症的发生密切相关。AtPRMT5是拟南芥中一个重要的蛋白质精氨酸甲基转移酶,能够催化组蛋白和非组蛋白的对称性双甲基化,AtPRMT5的缺失会导致植物生长发育的多种缺陷以及大量mRNA前体拼接异常,因此AtPRMT5通过调控植物生命周期各个阶段中mRNA前体的正确加工,保证了植物正常的生长发育过程。然而,对于AtPRMT5参与mRNA前体加工的分子机制的认识还非常有限。
中国科学院遗传与发育生物学研究所曹晓风研究组在前期研究的基础上,利用遗传学、转录组学、蛋白质组学和生物化学等研究手段,阐释了AtPRMT5参与mRNA前体加工的分子机制。曹晓风研究团队利用遗传筛选手段鉴定了两个能够部分回复atprmt5突变体生长发育异常表型以及mRNA前体拼接异常的抑制子,图位克隆显示这两个抑制子编码拼接复合体中U5 snRNP中的核心成员——Prp8 (分别为Prp8P1141S和Prp8P347S点突变)。进一步通过蛋白质组学研究发现,在atprmt5突变体中,拼接激活过程中的重要复合体Prp19 complex以及转酯反应特异的拼接因子与U5 snRNP之间的相互作用明显减弱,而在atprmt5抑制子中得到恢复。拼接复合体中U snRNP的核心组分Sm蛋白对称性双甲基化的缺失也会导致Prp19 complex与其相互作用的减弱,表明AtPRMT5通过影响其底物Sm蛋白与拼接复合体中其它蛋白之间的相互作用从而参与调控了拼接复合体的激活过程。
该项研究成功解析了AtPRMT5参与mRNA前体加工的分子机制,揭示了蛋白质精氨酸甲基转移酶参与高等植物复杂生命活动的调控机理,是 PRMT5参与拼接复合体组装机理研究方面的重要进展。该研究不仅对研究植物精氨酸甲基化调控生长发育的分子机理提供了重要的指导意义,同时也为研究人类遗传病和癌症的发生发展提供更多在转录后调控层级的线索和理论借鉴。
上述研究成果于2016年4月25日在PNAS上在线发表(doi: 10.1073/pnas.1522458113)。曹晓风研究组的助理研究员邓娴、博士后陆天聪和王璐璐为该论文的共同第一作者,曹晓风研究员和刘春艳副研究员为共同通讯作者。该研究得到了国家自然科学基金项目、科技部重大研究计划和植物基因组学国家重点实验室的资助。
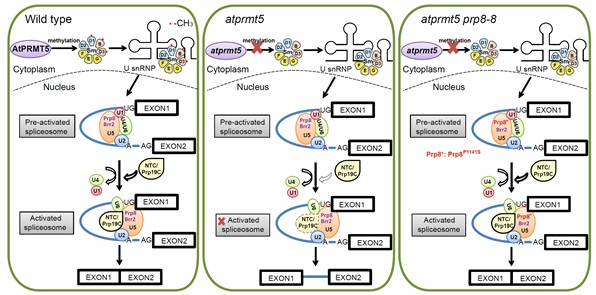
AtPRMT5参与mRNA前体加工的工作模型